Manipulación de datos ordenados usando {dplyr} - Primera parte
El paquete {dplyr} provee una enorme cantidad de funciones útiles para manipular y analizar datos de manera intuitiva y expresiva.
El espíritu detrás de {dplyr} es que la enorme mayoría de los análisis, por más complicados que sean, involucran combinar y encadenar una serie relativamente acotada de acciones (o verbos). Vamos a comenzar a trabajar con las cinco más comunes:
select(): selecciona columnas de una tabla.filter(): selecciona (o filtra) filas de una tabla a partir de una o más condiciones lógicas.group_by(): agrupa una tabla en base al valor de una o más columnas.mutate(): agrega nuevas columnas a una tabla.summarise(): calcula estadísticas para cada grupo de una tabla.
Primer desafío:
Te dieron una tabla con datos de temperatura mínima y máxima para
distintas estaciones meteorológicas de todo el país durante los 365 días
de un año. Las columnas son: id_estacion,
temperatura_maxima, temperatura_minima y
provincia. En base a esos datos, te piden que computen la
temperatura media anual de cada estación únicamente de las estaciones de
La Pampa.
¿En qué orden ejecutarías estos pasos para obtener el resultado deseado? Todavía sin correr ninguna línea de código.
- usar
summarise()para calcular la estadísticamean(temperatura_media)para cadaid_estacion - usar
group_by()para agrupar por la columnaid_estacion - usar
mutate()para agregar una columna llamadatemperatura_mediaque sea(temperatura_minima + temperatura_maxima)/2. - usar
filter()para seleccionar solo las filas donde la columnasprovinciaes igual a “La Pampa”
Para usar {dplyr} primero hay que instalarlo con el comando:
install.packages('dplyr')o junto con todos los paquetes del universo de Tidyverse y luego cargarlo en memoria con
library(dplyr)Cargar los datos de la estación de Bariloche (para un recordatorio, podés ir a Lectura de datos ordenados):
library(readr)
bariloche <- read_csv("datos/bariloche_enlimpio.csv")## Rows: 3024 Columns: 31
## ── Column specification ────────────────────────────────────────────────────────
## Delimiter: ","
## chr (3): Direccion_Viento_200cm, Direccion_Viento_1000cm, mes
## dbl (13): Temperatura_Abrigo_150cm, Temperatura_Abrigo_150cm_Maxima, Temper...
## lgl (14): Temperatura_Intemperie_5cm_Minima, Temperatura_Intemperie_50cm_Mi...
## dttm (1): Fecha
##
## ℹ Use `spec()` to retrieve the full column specification for this data.
## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.Seleccionando columnas con select()
Para quedarse únicamente con las columnas de Fecha,
Temperatura_Abrigo_150cm y Humedad_Media podés usar
select(), el primer argumento es siempre el data.frame:
select(bariloche, Fecha, Temperatura_Abrigo_150cm, Humedad_Media) # Ojo con las mayúsculas!## # A tibble: 3,024 × 3
## Fecha Temperatura_Abrigo_150cm Humedad_Media
## <dttm> <dbl> <dbl>
## 1 2011-05-28 00:00:00 NA NA
## 2 2011-05-29 00:00:00 4.27 79
## 3 2011-05-30 00:00:00 1.69 93
## 4 2011-05-31 00:00:00 3.47 96
## 5 2011-06-01 00:00:00 2.90 98
## 6 2011-06-02 00:00:00 6.26 93
## 7 2011-06-03 00:00:00 3.45 88
## 8 2011-06-04 00:00:00 6.09 87
## 9 2011-06-05 00:00:00 5.00 100
## 10 2011-06-06 00:00:00 5.45 91
## # … with 3,014 more rows¿Dónde quedó este resultado? Si te fijás en la variable
bariloche, ésta está intacta:
bariloche## # A tibble: 3,024 × 31
## Fecha Temperatura_Abrigo_150… Temperatura_Abr… Temperatura_Abr…
## <dttm> <dbl> <dbl> <dbl>
## 1 2011-05-28 00:00:00 NA NA NA
## 2 2011-05-29 00:00:00 4.27 12.8 -1.2
## 3 2011-05-30 00:00:00 1.69 9.2 -2.8
## 4 2011-05-31 00:00:00 3.47 9.6 0.5
## 5 2011-06-01 00:00:00 2.90 9.6 -1.5
## 6 2011-06-02 00:00:00 6.26 13.3 2.9
## 7 2011-06-03 00:00:00 3.45 12.6 -1.8
## 8 2011-06-04 00:00:00 6.09 10.2 3.9
## 9 2011-06-05 00:00:00 5.00 7.1 3.4
## 10 2011-06-06 00:00:00 5.45 9.5 3.2
## # … with 3,014 more rows, and 27 more variables:
## # Temperatura_Intemperie_5cm_Minima <lgl>,
## # Temperatura_Intemperie_50cm_Minima <lgl>,
## # Temperatura_Suelo_5cm_Media <lgl>, Temperatura_Suelo_10cm_Media <dbl>,
## # Temperatura_Inte_5cm <lgl>, Temperatura_Intemperie_150cm_Minima <lgl>,
## # Humedad_Suelo <lgl>, Precipitacion_Pluviometrica <dbl>,
## # Precipitacion_Cronologica <dbl>, Precipitacion_Maxima_30minutos <dbl>, …select() y el resto de las funciones de {dplyr} siempre
generan una nueva tabla y nunca modifican la tabla original. Para
guardar la tabla con las tres columnas Fecha,
Temperatura_Abrigo_150cm y Humedad_Media tenés
que asignar el resultado a una nueva variable.
temp_hum <- select(bariloche, Fecha, Temperatura_Abrigo_150cm, Humedad_Media)
temp_hum## # A tibble: 3,024 × 3
## Fecha Temperatura_Abrigo_150cm Humedad_Media
## <dttm> <dbl> <dbl>
## 1 2011-05-28 00:00:00 NA NA
## 2 2011-05-29 00:00:00 4.27 79
## 3 2011-05-30 00:00:00 1.69 93
## 4 2011-05-31 00:00:00 3.47 96
## 5 2011-06-01 00:00:00 2.90 98
## 6 2011-06-02 00:00:00 6.26 93
## 7 2011-06-03 00:00:00 3.45 88
## 8 2011-06-04 00:00:00 6.09 87
## 9 2011-06-05 00:00:00 5.00 100
## 10 2011-06-06 00:00:00 5.45 91
## # … with 3,014 more rows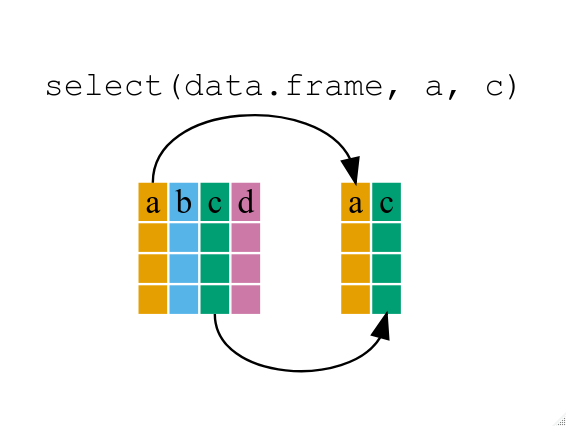
Cómo funciona select()
Filtrando filas con filter()
Ahora podés usar filter() para quedarte con sólo unas
filas. Por ejemplo, para ver aquellos días con temperatura menor a 0
grados:
filter(temp_hum, Temperatura_Abrigo_150cm < 0)## # A tibble: 38 × 3
## Fecha Temperatura_Abrigo_150cm Humedad_Media
## <dttm> <dbl> <dbl>
## 1 2011-06-25 00:00:00 -1.91 86
## 2 2011-07-02 00:00:00 -0.865 79
## 3 2011-07-03 00:00:00 -1.77 80
## 4 2011-07-04 00:00:00 -1.18 82
## 5 2011-07-05 00:00:00 -1.58 90
## 6 2011-07-17 00:00:00 -0.647 86
## 7 2011-07-18 00:00:00 -0.126 86
## 8 2011-07-31 00:00:00 -0.756 89
## 9 2012-06-06 00:00:00 -1.92 83
## 10 2012-06-07 00:00:00 -0.915 85
## # … with 28 more rowsLa mayoría de los análisis consisten en varios pasos (en el primer
desafío usaste 4 pasos para calcular las temperaturas medias de una
serie de estaciones). La única tabla que te interesa es la última, por
lo que ir asignando variables nuevas en cada paso intermedio es tedioso
y poco práctico. Para eso se usa el operador ‘pipe’
(%>%).
El operador ‘pipe’ (%>%) agarra el resultado de una
función y se lo pasa a la siguiente. Esto permite escribir el código
como una cadena de funciones que van operando sobre el resultado de la
anterior.
Las dos operaciones anteriores (seleccionar tres columnas y luego filtar las filas correspondientes temperaturas menores a 0°C) se pueden escribir uno después del otro y sin asignar los resultados intermedios a nuevas variables de esta forma:
bariloche %>%
select(Fecha, Temperatura_Abrigo_150cm, Humedad_Media) %>%
filter(Temperatura_Abrigo_150cm < 0)## # A tibble: 38 × 3
## Fecha Temperatura_Abrigo_150cm Humedad_Media
## <dttm> <dbl> <dbl>
## 1 2011-06-25 00:00:00 -1.91 86
## 2 2011-07-02 00:00:00 -0.865 79
## 3 2011-07-03 00:00:00 -1.77 80
## 4 2011-07-04 00:00:00 -1.18 82
## 5 2011-07-05 00:00:00 -1.58 90
## 6 2011-07-17 00:00:00 -0.647 86
## 7 2011-07-18 00:00:00 -0.126 86
## 8 2011-07-31 00:00:00 -0.756 89
## 9 2012-06-06 00:00:00 -1.92 83
## 10 2012-06-07 00:00:00 -0.915 85
## # … with 28 more rowsLa forma de “leer” esto es “Tomá la variable bariloche,
después aplicale
select(Fecha, Temperatura_Abrigo_150cm, Humedad_Media),
después aplicale
filter(Temperatura_Abrigo_150cm < 0)”.
Cómo vimos, el primero argumento de todas las funciones de dplyr
espera un data.frame y justamente el operador %>% toma
el data.frame bariloche y se lo pasa al primer argumento de
select(). Luego el data.frame resultante de seleccionar las
columnas Fecha, Temperatura_Abrigo_150cm y Humedad_Media pasa
como el primer argumento de la función filter() gracias al
%>%.
Tip:
En RStudio podés escribir %>% usando el atajo de
teclado Ctr + Shift + M. ¡Probalo!
Desafío:
Completá esta cadena para producir una tabla que contenga los valores de Fecha, Temperatura del suelo y la Humedad únicamente cuando la Humedad es igual a 100%.
bariloche %>%
filter(Humedad_Media == ___) %>%
select(___, ___, ___)Agrupando y reduciendo con
group_by() %>% summarise()
Si querés calcular la temperatura media para cada mes, tenés que usar
el combo group_by() %>% summarise(). Es decir, primero
agrupar la tabla según la columna mes y luego calcular un
promedio de Temperatura_Abrigo_150cm para cada grupo.
Para agrupar la tabla bariloche según el mes usamos el
siguiente código:
bariloche %>%
group_by(mes) ## # A tibble: 3,024 × 31
## # Groups: mes [12]
## Fecha Temperatura_Abrigo_150… Temperatura_Abr… Temperatura_Abr…
## <dttm> <dbl> <dbl> <dbl>
## 1 2011-05-28 00:00:00 NA NA NA
## 2 2011-05-29 00:00:00 4.27 12.8 -1.2
## 3 2011-05-30 00:00:00 1.69 9.2 -2.8
## 4 2011-05-31 00:00:00 3.47 9.6 0.5
## 5 2011-06-01 00:00:00 2.90 9.6 -1.5
## 6 2011-06-02 00:00:00 6.26 13.3 2.9
## 7 2011-06-03 00:00:00 3.45 12.6 -1.8
## 8 2011-06-04 00:00:00 6.09 10.2 3.9
## 9 2011-06-05 00:00:00 5.00 7.1 3.4
## 10 2011-06-06 00:00:00 5.45 9.5 3.2
## # … with 3,014 more rows, and 27 more variables:
## # Temperatura_Intemperie_5cm_Minima <lgl>,
## # Temperatura_Intemperie_50cm_Minima <lgl>,
## # Temperatura_Suelo_5cm_Media <lgl>, Temperatura_Suelo_10cm_Media <dbl>,
## # Temperatura_Inte_5cm <lgl>, Temperatura_Intemperie_150cm_Minima <lgl>,
## # Humedad_Suelo <lgl>, Precipitacion_Pluviometrica <dbl>,
## # Precipitacion_Cronologica <dbl>, Precipitacion_Maxima_30minutos <dbl>, …A primera vista parecería que la función no hizo nada, pero fijate que el resultado ahora dice que tiene grupos (“Groups:”), y nos dice qué columna es la que agrupa los datos (“mes”) y cuántos grupos hay (“12”). Las operaciones subsiguientes que le hagamos a esta tabla van a hacerse para cada grupo.
Para ver esto en acción, usá summarise() para computar
el promedio de la temperatura:
bariloche %>%
group_by(mes) %>%
summarise(Temperatura_media = mean(Temperatura_Abrigo_150cm))## # A tibble: 12 × 2
## mes Temperatura_media
## <chr> <dbl>
## 1 abril NA
## 2 agosto NA
## 3 diciembre 14.0
## 4 enero NA
## 5 febrero NA
## 6 julio NA
## 7 junio NA
## 8 marzo NA
## 9 mayo NA
## 10 noviembre NA
## 11 octubre NA
## 12 septiembre NA¡Tadá! summarise() devuelve una tabla con una columna
para el continente y otra nueva, llamada “Temperatura_media” que
contiene el promedio de Temperatura_Abrigo_150cm para cada
grupo. Pero… aparecen muchos datos faltantes, cuando en realidad hay
muchas observaciones para cada mes.
Si revisamos la ayuda de la función mean(), vemos que
tiene un argumento que por defecto es na.rm = FALSE, esto
significa que si al menos una observación es NA, el
resultado será NA. Cambiemos este argumento a
TRUE para cambiar el comportamiento de la función.
bariloche %>%
group_by(mes) %>%
summarise(Temperatura_media = mean(Temperatura_Abrigo_150cm, na.rm = TRUE))## # A tibble: 12 × 2
## mes Temperatura_media
## <chr> <dbl>
## 1 abril 10.5
## 2 agosto 4.83
## 3 diciembre 14.0
## 4 enero 16.6
## 5 febrero 16.0
## 6 julio 3.69
## 7 junio 5.18
## 8 marzo 13.8
## 9 mayo 7.34
## 10 noviembre 11.6
## 11 octubre 8.59
## 12 septiembre 6.43Ahora si, tenemos la temperatura media para cada mes.
group_by() permite agrupar en base a múltiples columnas y
summarise() permite generar múltiples columnas de resumen.
El resultado va a siempre ser una tabla con la misma cantidad de filas
que grupos y una cantidad de columnas igual a la cantidad de columnas
usadas para agrupar y los estadísticos computados.
Desafío:
¿Cuál te imaginás que va a ser el resultado del siguiente código? ¿Cuántas filas y columnas va a tener? (Tratá de pensarlo antes de correrlo.)
bariloche %>%
summarise(Temperatura_media = mean(Temperatura_Abrigo_150cm))El combo group_by() %>% summarise() se puede resumir
en esta figura. Las filas de una tabla se dividen en grupos, y luego
cada grupo se “resume” en una fila en función del estadístico usado.
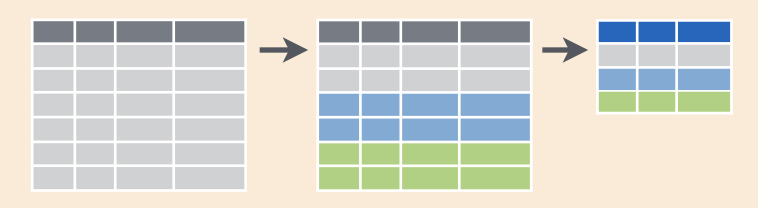
Al igual que hicimos “cuentas” usando algunas variables numéricas
para obtener información nueva, también podemos utilizar variables
categóricas. No tiene sentido calcular mean(mes) ya que
contienen caracteres, pero tal vez nos interese contar la
cantidad de observaciones por mes:
bariloche %>%
group_by(mes) %>%
summarise(cantidad = n())## # A tibble: 12 × 2
## mes cantidad
## <chr> <int>
## 1 abril 239
## 2 agosto 276
## 3 diciembre 248
## 4 enero 248
## 5 febrero 227
## 6 julio 273
## 7 junio 254
## 8 marzo 248
## 9 mayo 239
## 10 noviembre 241
## 11 octubre 279
## 12 septiembre 252En R se puede resolver un problema de muchas maneras, por ejemplo, el código anterior y el que sigue dan resultados equivalentes:
bariloche %>%
group_by(mes) %>%
count()## # A tibble: 12 × 2
## # Groups: mes [12]
## mes n
## <chr> <int>
## 1 abril 239
## 2 agosto 276
## 3 diciembre 248
## 4 enero 248
## 5 febrero 227
## 6 julio 273
## 7 junio 254
## 8 marzo 248
## 9 mayo 239
## 10 noviembre 241
## 11 octubre 279
## 12 septiembre 252En este caso count() es análogo a
summarise(cantidad = n()).
Creando nuevas columnas con mutate()
Todo esto está bien para hacer cálculos con columnas previamente existentes, pero muchas veces tenés que crear nuevas columnas.
La tabla bariloche tiene información de temperaturas en
grados centígrados y puede ser necesario convertirlas a kelvin.
mutate() permite agregar una nueva columna llamada
“Temperatura_kelvin” con esa información:
bariloche %>%
mutate(Temperatura_kelvin = Temperatura_Abrigo_150cm + 273,15)## # A tibble: 3,024 × 33
## Fecha Temperatura_Abrigo_150… Temperatura_Abr… Temperatura_Abr…
## <dttm> <dbl> <dbl> <dbl>
## 1 2011-05-28 00:00:00 NA NA NA
## 2 2011-05-29 00:00:00 4.27 12.8 -1.2
## 3 2011-05-30 00:00:00 1.69 9.2 -2.8
## 4 2011-05-31 00:00:00 3.47 9.6 0.5
## 5 2011-06-01 00:00:00 2.90 9.6 -1.5
## 6 2011-06-02 00:00:00 6.26 13.3 2.9
## 7 2011-06-03 00:00:00 3.45 12.6 -1.8
## 8 2011-06-04 00:00:00 6.09 10.2 3.9
## 9 2011-06-05 00:00:00 5.00 7.1 3.4
## 10 2011-06-06 00:00:00 5.45 9.5 3.2
## # … with 3,014 more rows, and 29 more variables:
## # Temperatura_Intemperie_5cm_Minima <lgl>,
## # Temperatura_Intemperie_50cm_Minima <lgl>,
## # Temperatura_Suelo_5cm_Media <lgl>, Temperatura_Suelo_10cm_Media <dbl>,
## # Temperatura_Inte_5cm <lgl>, Temperatura_Intemperie_150cm_Minima <lgl>,
## # Humedad_Suelo <lgl>, Precipitacion_Pluviometrica <dbl>,
## # Precipitacion_Cronologica <dbl>, Precipitacion_Maxima_30minutos <dbl>, …Recordá que las funciones de {dplyr} nunca modifican la tabla
original. mutate() devolvió una nueva tabla que es igual a
la tabla bariloche pero con la columna “Temperatura_kelvin”
agregada. La tabla bariloche sigue intacta.
Si quisiéramos aplicar la misma operación a todas las columnas que
tienen temperatura, la función across() brinda un atajo
para no tener que aplicar mutate() una y otra vez a cada
cada columna.
bariloche %>%
summarise(across(starts_with("Temperatura"), ~.x + 273,15))## # A tibble: 3,024 × 9
## Temperatura_Abrigo_150cm Temperatura_Abrig… Temperatura_Abr… Temperatura_Int…
## <dbl> <dbl> <dbl> <dbl>
## 1 NA NA NA NA
## 2 277. 286. 272. NA
## 3 275. 282. 270. NA
## 4 276. 283. 274. NA
## 5 276. 283. 272. NA
## 6 279. 286. 276. NA
## 7 276. 286. 271. NA
## 8 279. 283. 277. NA
## 9 278. 280. 276. NA
## 10 278. 282. 276. NA
## # … with 3,014 more rows, and 5 more variables:
## # Temperatura_Intemperie_50cm_Minima <dbl>,
## # Temperatura_Suelo_5cm_Media <dbl>, Temperatura_Suelo_10cm_Media <dbl>,
## # Temperatura_Inte_5cm <dbl>, Temperatura_Intemperie_150cm_Minima <dbl>Varias cosas pasaron acá:
- El resultado solo incluye las columnas asociadas a la temperatura.
- No fue necesario escribir el nombre de cada columna, la función
start_with()automáticamente busca las columnas que comienzan con la palabra que indicamos, en este caso “Temperatura”. - Usamos la notación funcional de R, que arranca con
~y luego le indicamos que a .x (cada columna) le sume 273,15.
Desafío
Si quisieras calcular la temperatura media para cada día y guardarla en una nueva variable, ¿cómo completarías el siguiente código?
bariloche %>%
mutate(____ = ____)Ahora imaginemos que necesitamos modificar algunas observaciones, aquellas que cumplen con una condición. Por ejemplo, descubrimos que cuando la humedad es 100%, el sensor de temperatura no funciona correctamente y necesitamos aplicarle una corrección de -2° en esas situaciones.
Podríamos filtrar las observaciones que cumplen con que
Humedad_Media == 100 y luego modificar la temperatura, pero
entonces tendríamos una base de datos para las observaciones con mayor
humedad y la temperatura corregida y otra base de datos sin la
corrección. Necesitamos poder modificar solo algunas
observaciones y que el resto se mantenga igual.
Para eso podemos utilizar la función if_else(), que
aplica una operación si ocurre algo y otra operación si
no. En este caso si Humedad_Media == 100 es verdadera,
restamos 2 grados a la Temperatura, si es falsa no la modificamos. Y por
supuesto se la aplicamos a la columna con mutate()
bariloche %>%
mutate(Temperatura_Abrigo_150cm = if_else(Humedad_Media == 100, # Condición
Temperatura_Abrigo_150cm -2,# Si es verdadera
Temperatura_Abrigo_150cm)) # Si es falsa## # A tibble: 3,024 × 31
## Fecha Temperatura_Abrigo_150… Temperatura_Abr… Temperatura_Abr…
## <dttm> <dbl> <dbl> <dbl>
## 1 2011-05-28 00:00:00 NA NA NA
## 2 2011-05-29 00:00:00 4.27 12.8 -1.2
## 3 2011-05-30 00:00:00 1.69 9.2 -2.8
## 4 2011-05-31 00:00:00 3.47 9.6 0.5
## 5 2011-06-01 00:00:00 2.90 9.6 -1.5
## 6 2011-06-02 00:00:00 6.26 13.3 2.9
## 7 2011-06-03 00:00:00 3.45 12.6 -1.8
## 8 2011-06-04 00:00:00 6.09 10.2 3.9
## 9 2011-06-05 00:00:00 3.00 7.1 3.4
## 10 2011-06-06 00:00:00 5.45 9.5 3.2
## # … with 3,014 more rows, and 27 more variables:
## # Temperatura_Intemperie_5cm_Minima <lgl>,
## # Temperatura_Intemperie_50cm_Minima <lgl>,
## # Temperatura_Suelo_5cm_Media <lgl>, Temperatura_Suelo_10cm_Media <dbl>,
## # Temperatura_Inte_5cm <lgl>, Temperatura_Intemperie_150cm_Minima <lgl>,
## # Humedad_Suelo <lgl>, Precipitacion_Pluviometrica <dbl>,
## # Precipitacion_Cronologica <dbl>, Precipitacion_Maxima_30minutos <dbl>, …La mayoría de las funciones de R pueden trabajar sobre vectores (y las columnas de un data.frame son como vectores ordenados!) y lo hacen elemento a elemento.
En este caso if_else() recorre la columna con la
información de humedad y elemento a elemento revisa si el valor
es igual a 100. Luego decide si debe modificar o no cada elemento, en el
orden en el que aparecen.
Desafío
Ahora es tu turno, “arreglá” las variables
Temperatura_Abrigo_150cm_Maxima y
Temperatura_Abrigo_150cm_Minima para aplicar la misma
corrección de 2 grados.
Desagrupando con ungroup()
En general, la mayoría de las funciones de {dplyr} “entienden” cuando una tabla está agrupada y realizan las operaciones para cada grupo.
Desafío:
¿Cuál de estos dos códigos agrega una columna llamada “Temperatura_max_media” con el la temperatura máxima media para cada mes? ¿Qué hace el otro?
bariloche %>%
group_by(mes) %>%
mutate(Temperatura_max_media = mean(Temperatura_Abrigo_150cm_Maxima, na.rm = TRUE))
bariloche %>%
mutate(Temperatura_max_media = mean(Temperatura_Abrigo_150cm_Maxima, na.rm = TRUE)) En otras palabras, los resultados de mutate(),
filter(), summarise() y otras funciones
cambian según si la tabla está agrupada o no. Como a veces uno se puede
olvidar que quedaron grupos, es conveniente usar la función
ungroup() una vez que dejás de trabajar con grupos:
bariloche %>%
group_by(mes) %>%
mutate(Temperatura_max_media = mean(Temperatura_Abrigo_150cm_Maxima, na.rm = TRUE)) %>%
ungroup()## # A tibble: 3,024 × 32
## Fecha Temperatura_Abrigo_150… Temperatura_Abr… Temperatura_Abr…
## <dttm> <dbl> <dbl> <dbl>
## 1 2011-05-28 00:00:00 NA NA NA
## 2 2011-05-29 00:00:00 4.27 12.8 -1.2
## 3 2011-05-30 00:00:00 1.69 9.2 -2.8
## 4 2011-05-31 00:00:00 3.47 9.6 0.5
## 5 2011-06-01 00:00:00 2.90 9.6 -1.5
## 6 2011-06-02 00:00:00 6.26 13.3 2.9
## 7 2011-06-03 00:00:00 3.45 12.6 -1.8
## 8 2011-06-04 00:00:00 6.09 10.2 3.9
## 9 2011-06-05 00:00:00 5.00 7.1 3.4
## 10 2011-06-06 00:00:00 5.45 9.5 3.2
## # … with 3,014 more rows, and 28 more variables:
## # Temperatura_Intemperie_5cm_Minima <lgl>,
## # Temperatura_Intemperie_50cm_Minima <lgl>,
## # Temperatura_Suelo_5cm_Media <lgl>, Temperatura_Suelo_10cm_Media <dbl>,
## # Temperatura_Inte_5cm <lgl>, Temperatura_Intemperie_150cm_Minima <lgl>,
## # Humedad_Suelo <lgl>, Precipitacion_Pluviometrica <dbl>,
## # Precipitacion_Cronologica <dbl>, Precipitacion_Maxima_30minutos <dbl>, …