Graficando
Ahora que tenés los datos en forma de tabla, todo lo que queda es usar las mismas herramientas que usarías para trabajar con cualquier otro tipo de dato.
# Cargo los paquetes necesarios
library(magrittr)
library(ggplot2)
library(dplyr)
library(data.table)
library(metR)
# Leo los datos
#
sst <- ReadNetCDF("datos/temperatura_mar.nc", vars = "sst")
# Me quedo con un solo campo, para graficar
sst1 <- sst[time == time[1]]
sst1 <- sst %>%
filter(time == time[1])Ya vimos una forma rápida de graficar este tipo de datos usando
geom_raster():
sst1 %>%
ggplot(aes(longitude, latitude)) +
geom_raster(aes(fill = sst)) 
Con geom_raster(), cada punto de grilla es representado
como un rectángulo cuyo color de relleno se mapea al valor de la
variable, por lo tanto la estética a usar es fill.
Contornos
Otra forma de mostrar este tipo de campos (o sea, que pinta tiene la
variable en una región) es usando contornos con
geom_contour(). A mí me gusta usar
metR::geom_contour2() porque usa contornos negros por
default y tiene otras funcionalidades útiles. En este caso, cada línea
corresponde a un valor específico de la variable y por eso usamos el
argumento z para mapearla.
sst1 %>%
ggplot(aes(longitude, latitude)) +
geom_contour2(aes(z = sst)) 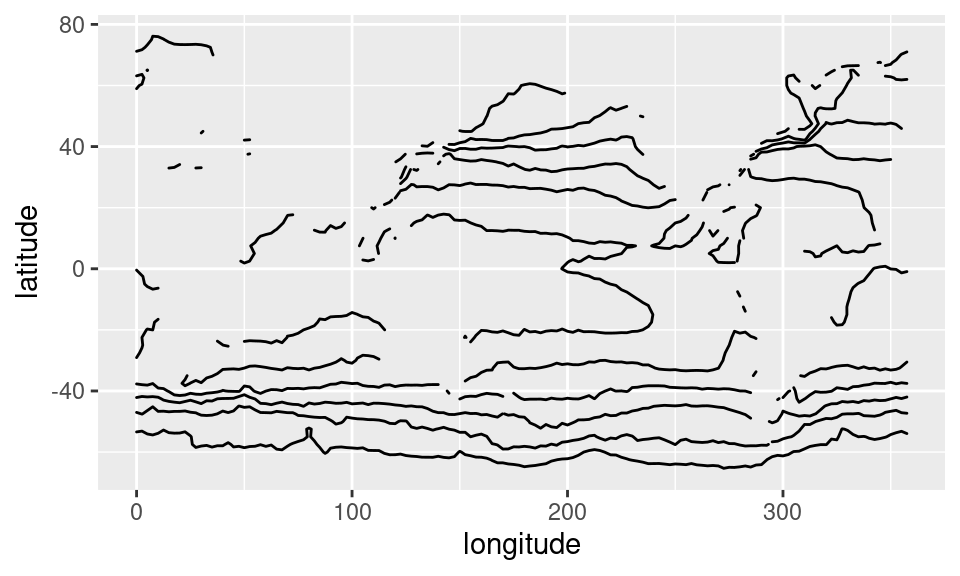
El problema es que ahora perdemos los continentes gratis y no tenemos
información sobre que valores representa cada línea. Podemos obtener un
resultado intermedio usando contornos llenos con
metR::geom_contour_fill() (de nuevo, ggplot2 tiene un
geom_contour_filled() pero yo prefiero los defaults y otras
funcionalidades de geom_contour_fill()).
sst1 %>%
ggplot(aes(longitude, latitude)) +
geom_contour_fill(aes(z = sst)) 
En este punto estaría bueno tener no tener los continentes pixelados
(esto se debe a la baja resolución de los datos principalmente). El
paquete rnaturalearth tiene datos de costas, países y
regiones en distintas resoluciones. Pero por ahora podemos evitar
instalar otro paquete, usando la función map_data() de
ggplot2.
ggplot() +
geom_polygon(data = map_data("world2"), aes(long, lat, group = group),
fill = "white")
Ahora podemos poner eso encima del gráfico anterior.
mapa <- geom_polygon(data = map_data("world2"), aes(long, lat, group = group),
fill = "white", inherit.aes = FALSE)sst1 %>%
ggplot(aes(longitude, latitude)) +
geom_contour_fill(aes(z = sst)) +
mapa
Aún con esto seguimos viendo las cosas “pixeladas”. Una forma de
resolver esto es rellenando las regiones sin datos con un valor
razonable. metR::geom_contour_fill() tiene el argumento
na.fill que si es TRUE, interpola los datos
faltantes:
sst1 %>%
ggplot(aes(longitude, latitude)) +
geom_contour_fill(aes(z = sst), na.fill = TRUE) 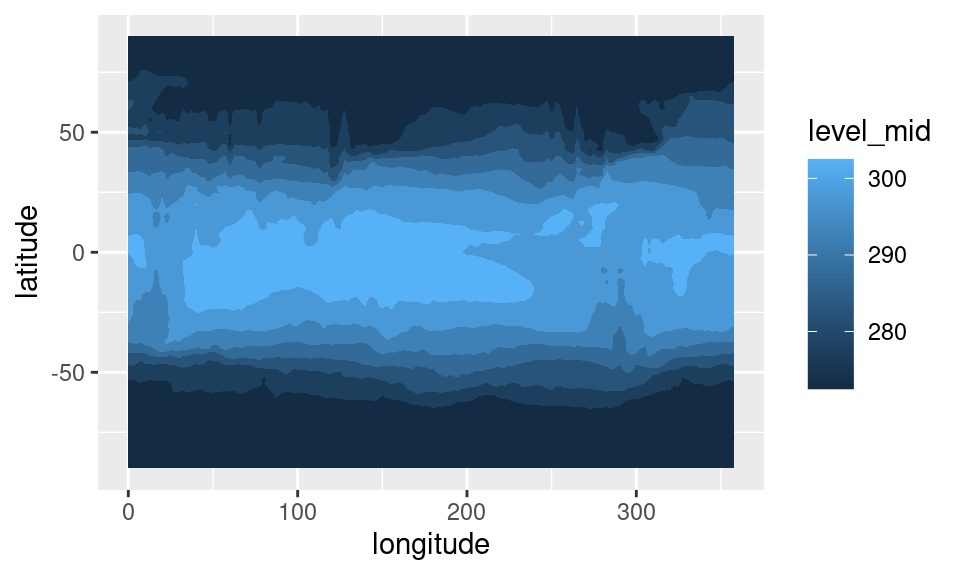
Sabemos que los datos bien adentro de los continentes no tienen sentido, pero si lo que vamos a hacer es taparlos con el mapa, no tiene mucha importancia.
sst1 %>%
ggplot(aes(longitude, latitude)) +
geom_contour_fill(aes(z = sst), na.fill = TRUE) +
mapa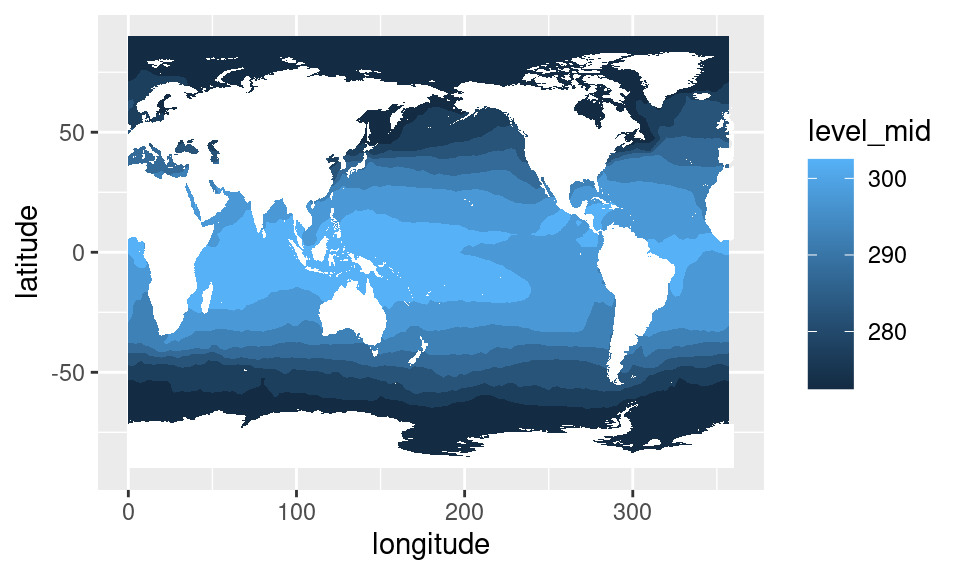
Usando proyecciones
Hasta ahora usamos datos grillados en una grilla regular en longitud
y latitud, pero no todos los datos son así. Por ejemplo el dataset de
ejemplo surface tiene datos de topografía del centro de
Argentina.
head(surface)## lon lat height x y
## 1: -74.83386 -40.91083 0.0000 -960000 -1160000
## 2: -74.36743 -40.94587 0.0000 -920000 -1160000
## 3: -73.90057 -40.97941 276.4575 -880000 -1160000
## 4: -73.43341 -41.01149 182.4261 -840000 -1160000
## 5: -72.96585 -41.04207 123.7324 -800000 -1160000
## 6: -72.49799 -41.07118 770.1770 -760000 -1160000Vemos que tiene datos de topografía en longitud y latitud, en este espacio los datos no son regulares ya que tiene en cuenta la curvatura de la Tierra. Pero además en el data.frame hay dos columnas x e y que corresponde a la distancia en metros entre las observaciones o datos al punto central de la proyección de estos datos. Vamos a ver qué pinta tienen cuando graficamos en función de la latitud y la longitud:
surface %>%
ggplot(aes(lon, lat)) +
geom_point(aes(colour = height))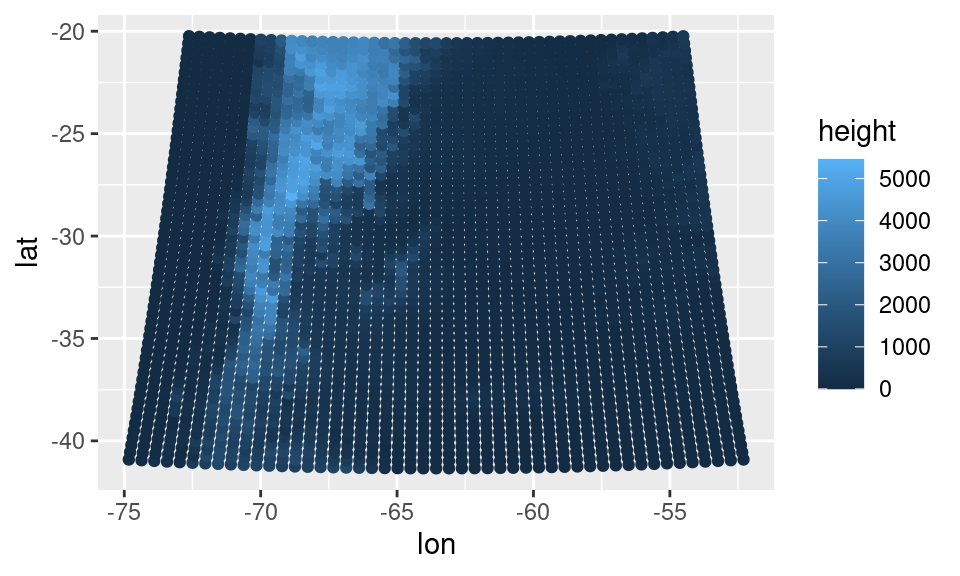
Claramente esto no es un cuadrado, pero hay una cierta regularidad en la ubicación de los puntos. La grilla sí es regular, pero en la proyección de Lambert. Si graficamos usando la información en x e y (o sea los datos “desproyectados”) nos encontramos con lo siguiente:
surface %>%
ggplot(aes(x, y)) +
geom_raster(aes(fill = height))
En esta grilla regular si podemos graficar contornos sin problema.
surface %>%
ggplot(aes(x, y)) +
geom_contour_fill(aes(z = height))
Una forma de definir esa proyección es con una “proj-string”; un texto que define de qué proyección se trata y cuáles son sus parámetros. (Aunque notar que este paradigma ya se quedó viejo) ¿De donde sale esta string? ¡De los metadatos! Si tu archivo netCDF está bien hecho, parte de sus metadatos va a ser la proyección. En este caso, el “metadato” es este documento y la proj-string es la siguiente:
proj_string <- paste0("+proj=lcc +lat_1=-30.9659996032715 +lat_2=-30.9659996032715 +lat_0=-30.9660034179688 +lon_0=296.432998657227 +a=6370000 +b=6370000 +over")Si incluimos esta información en argumento proj de
geom_contour_fill() podemos pasar de las coordenadas
proyectadas x/y a lon/lat y visualizar la región de manera correcta.
surface %>%
ggplot(aes(x, y)) +
geom_contour_fill(aes(z = height), proj = proj_string)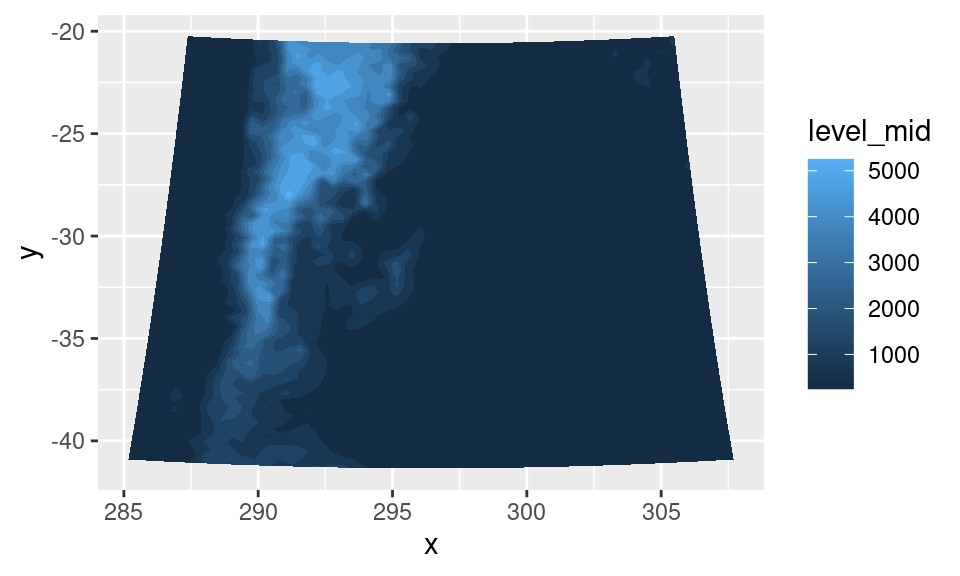
Graficando más de una variable
Muchas veces (al menos en meteorología!) queremos comparar varias variables al mismo tiempo y haces 2 gráficos no suele ser suficiente, queremos superponer las variables. ggplot2 tiene algunas limitaciones no podemos mapear el color a 2 variables distintas, al menos no directamente.
Una solución sería usar contornos vacíos y etiquetas para identificar valores (o aplicando color) sobre contornos llenos que usa el relleno o fill para definir los valores. De esa manera tendremos leyendas distintas (color -o etiquetas- y fill) para analizar las 2 variables.
Otra forma de plotear varias escalas de colores en un mismo ggplot es usando el paquete ggnewscale. En este ejemplo estamos graficando la topografía de la superficie de los continentes y del fondo del mar. Si bien podríamos trabajar con los datos como una única variable, suele ser útil diferenciarla.
Primero graficamos los datos donde h (la altura) es positiva (los
continentes) y luego agregamos ggnewscale::new_scale_fill()
para habilitar una nueva escala de relleno y usarla para los
datos con valor negativo (el fondo del océano).
topo <- GetTopography(270, 320, -20, -60, resolution = 1/30)
topo %>%
ggplot(aes(lon, lat)) +
geom_contour_fill(aes(z = h), data = ~.x[h > 0]) +
scale_fill_gradientn(colours = terrain.colors(4)) +
ggnewscale::new_scale_fill() +
geom_contour_fill(aes(z = h), data = ~.x[h <= 0]) 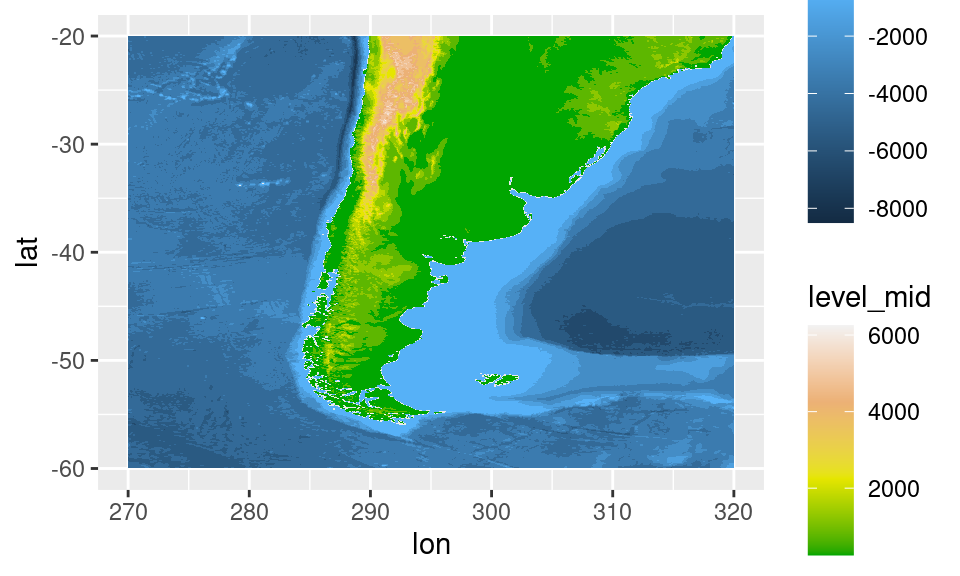
Guía discretizada
Un último truco que hace ayuda a interpretar los contornos llenos es
cambiar la guía de colores. geom_contour_fill() por defecto
usa una escala continua para el aes fill, por lo que la guía de colores
es un degradé continuo. El problema de eso es que no respeta
necesariamente la idea de que geom_contour_fill() en cierta
forma está discretizando los datos.
Una primera solución es usar una variable discreta. Para esto hay que
cambiar el aes fill a ..level.., que es la variable “level”
computada por el geom. Esta es una versión discreta de la variable usada
por default (..level_mid..), lo cual fuerza a gplot2 a
elegir una escala discreta junto con su guía.
sst1 %>%
.[longitude %between% c(270, 320) & latitude %between% c(-60, -20)] %>%
ggplot(aes(longitude, latitude)) +
geom_contour_fill(aes(z = sst, fill = ..level..), na.fill = TRUE) +
mapa +
coord_quickmap(xlim = c(270, 320), ylim = c(-60, -20)) 
El problema con esta escala es que no reconoce que si bien los datos ahora son discretos, ¡éstos representan números continuos! Para ilustrar el problema, veamos qué pasa si usamos breaks que no son equidistantes.
sst1 %>%
.[longitude %between% c(270, 320) & latitude %between% c(-60, -20)] %>%
ggplot(aes(longitude, latitude)) +
geom_contour_fill(aes(z = sst, fill = ..level..), na.fill = TRUE,
breaks= c(272, 275, 280, 295, 300)) +
mapa +
coord_quickmap(xlim = c(270, 320), ylim = c(-60, -20)) 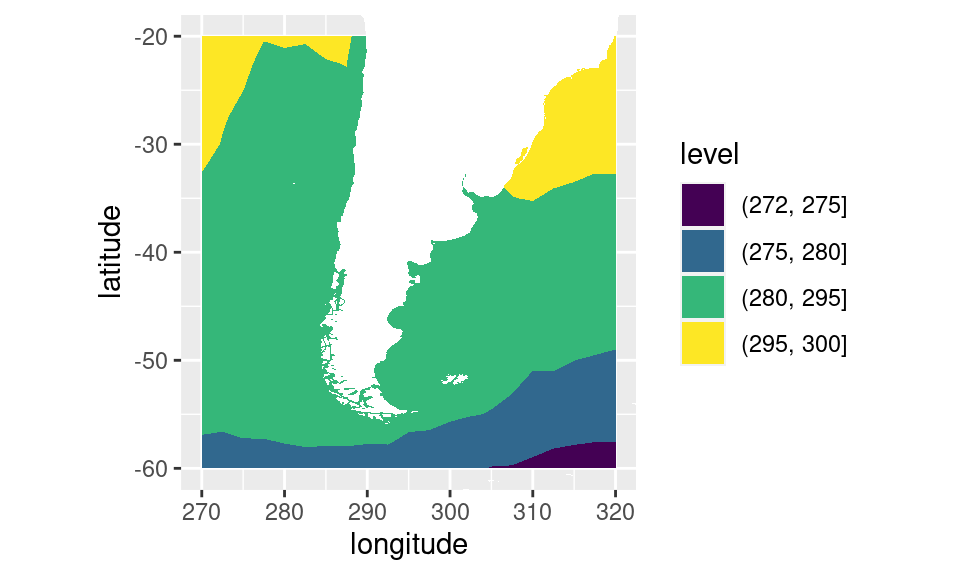
Como la escala es discreta, no tiene forma de interpretar que el “ancho” de la región entre 272 y 275 es muy distinto que el ancho entre 280 y 295. Esto se puede remediar usando una escala discretizada.
Cualquier escala contínua se puede convertir en una escala
discretizada agregando super = metR::ScaleDiscretised (el
metR:: no es necesario si tenés metR cargado, pero está acá
para indicar en de qué paquete sale). Además, hay que usar la escala
colorsteps de ggplot2 con el argumento
even.steps = FALSE.
sst1 %>%
.[longitude %between% c(270, 320) & latitude %between% c(-60, -20)] %>%
ggplot(aes(longitude, latitude)) +
geom_contour_fill(aes(z = sst, fill = ..level..), na.fill = TRUE,
breaks= c(272, 275, 280, 295, 300)) +
scale_fill_viridis_c(option = "B", super = metR::ScaleDiscretised,
guide = guide_colorsteps(even.steps = FALSE)) +
mapa +
coord_quickmap(xlim = c(270, 320), ylim = c(-60, -20)) 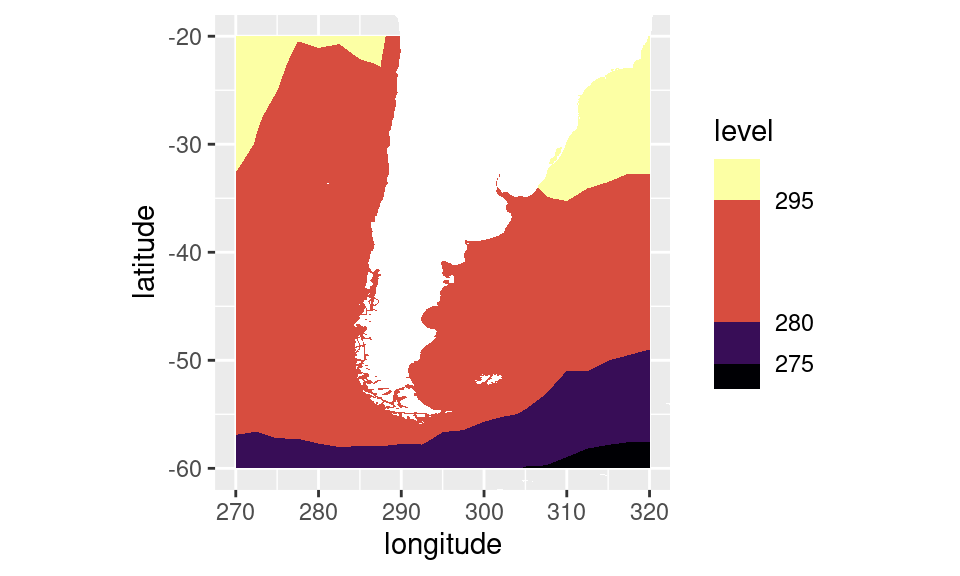
Contornos iluminados
Muchas veces los datos tienen un punto medio lógico y interesa ver
las desviaciones con respecto a ese punto medio. En ese caso, es muy
útil usar una escala de colores divergente. Para esto se puede usar
scale_fill_gradient2() o
metR::scale_fill_divergent() (que es lo mismo pero con el
default razonable de que valores mayores al punto medio están en rojo y
valores menores se pintan de azul), o la versión discretizada
metR::scale_fill_divergent_discretised().
sst1 %>%
ggplot(aes(longitude, latitude)) +
geom_contour_fill(aes(z = sst, fill = ..level..), na.fill = TRUE) +
scale_fill_divergent_discretised(midpoint = 290) +
mapa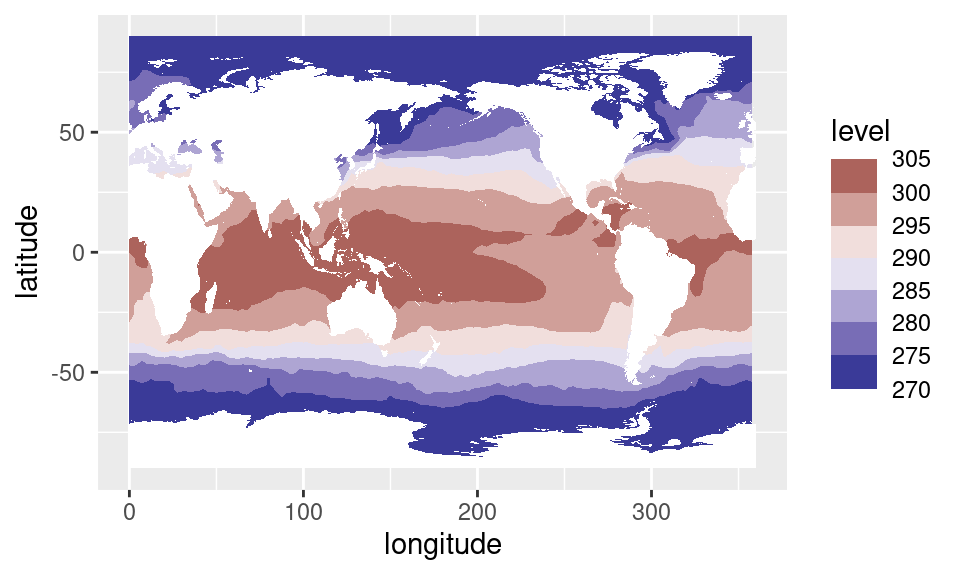
¿Pero qué pasa si este gráfico alguien lo veo imprime en blanco y negro? Lo que pasa es que los valores mayores al punto medio se ven iguales que los valores menores al punto medio.
colorblindr::edit_colors(last_plot(), colfun = colorspace::desaturate) %>%
cowplot::plot_grid()
Una posible solución es agregarle un efecto de relieve a los
contornos usando metR::geom_contour_tanaka().
sst1 %>%
ggplot(aes(longitude, latitude)) +
geom_contour_fill(aes(z = sst, fill = ..level..), na.fill = TRUE) +
geom_contour_tanaka(aes(z = sst), na.fill = TRUE) +
scale_fill_divergent_discretised(midpoint = 290) +
mapa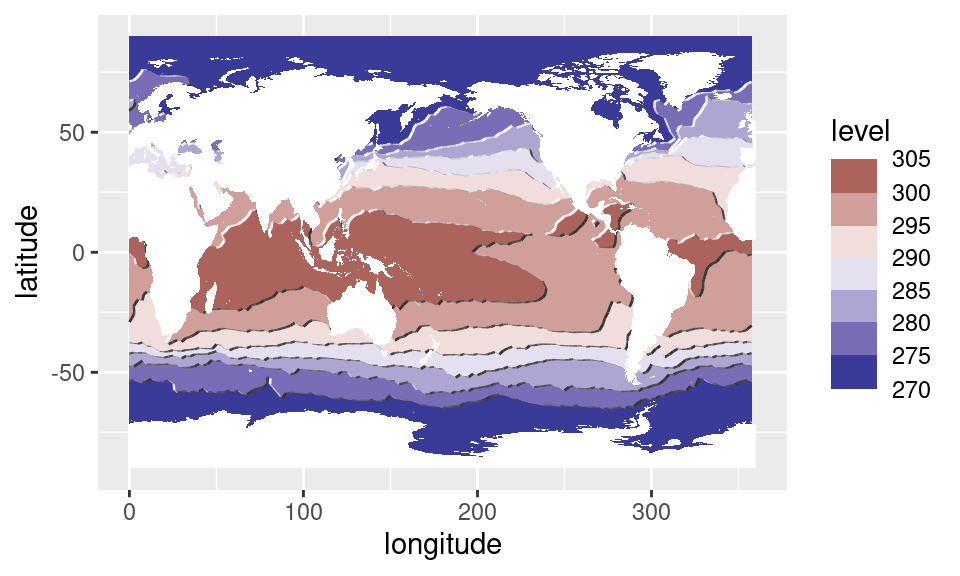
Además de quedar bonitos, ahora es muy fácil saber si un detarminado valor es una anomalía positiva o negativa aún en blanco y negro.
colorblindr::edit_colors(last_plot(), colfun = colorspace::desaturate) %>%
cowplot::plot_grid()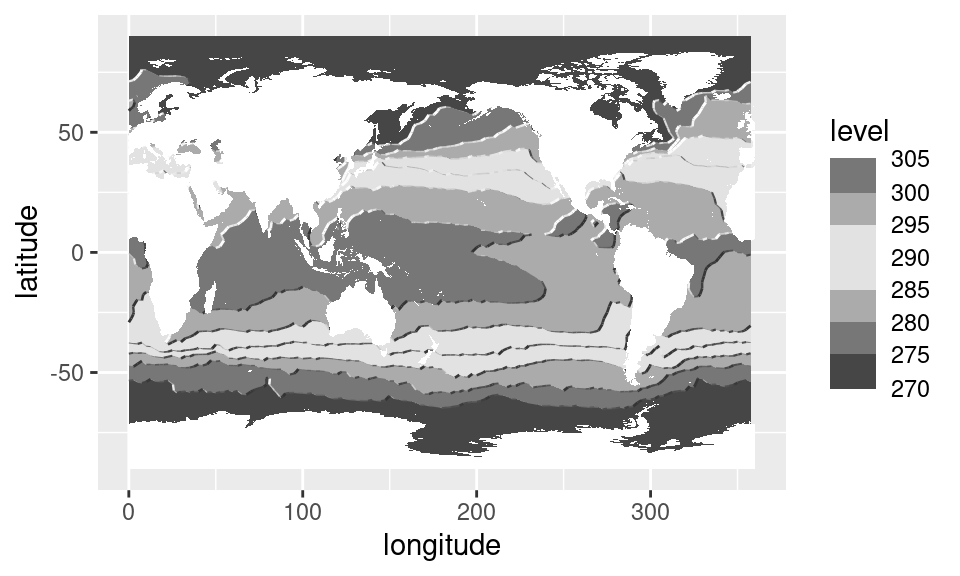
El paquete colorblindr es muy útil para ver que
pinta tiene el gráfico según distintos tipos de ceguera del color. Podés
instalarlo con
remotes::install_github("clauswilke/colorblindr")